fastqc命令说明,fastload命令
作者:admin 发布时间:2024-10-17 02:36 分类:资讯 浏览:3
今天给各位分享fastqc命令说明的知识,其中也会对fastload命令进行解释,如果能碰巧解决你现在面临的问题,别忘了关注本站,现在开始吧!
本文目录一览:
- 1、ATAC-seq专题---生信分析流程
- 2、二代测序的数据的分析——质量控制
- 3、3、RNAseq(3)--对RNAseq测序数据的质量控制(fastqc)
- 4、ChIP-seq数据质控与过滤
- 5、Trim_galore使用(2018-05-25)
- 6、RNA-Seq数据分析——原始数据质量控制(QC)
ATAC-seq专题---生信分析流程
1、ChIP-Seq原理是:首先通过染色质免疫共沉淀技术(ChIP) 特异性地富集目的蛋白结合的DNA片段 ,并对其进行纯化与文库构建;然后对富集得到的DNA片段进行高通量测序。ATAC-seq是 全基因组范围 内,找出所有的OCR。
2、相比起来,ATAC-seq的重复性,比MNase-seq和DNase-seq的更强,操作起来也更加简单,而且只需要很少的细胞/组织量,同时出来的信号更加漂亮。目前已经是研究染色质开放性首选的技术方法。
3、使用DiffBind包提取ATAC-seq数据中的差异peaks主要包括以下几个步骤:准备输入数据:首先,需要准备ATAC-seq数据,这通常是peak调用软件(如MACS2)产生的peak文件。对于多个样本,你需要为每个样本准备一个peak文件。
4、与单细胞转录组类似,单细胞ATAC的分析流程也主要包括细胞质控、peaks标准化及其降维分群、marker基因的鉴定等几个步骤。常用的单细胞ATAC分析流程软件包含 cell-ranger-atac、Signac和ArchR等。
5、首先,运行 cellranger-atac mkfastq 生成拆库后的fastq文件; 2)接下来,对于每个测序文库,识别相应的测序文件,其中R1和R3是测序的reads,I1是16bp i5 (10x Barcode), R2是i7 (sample index)。
二代测序的数据的分析——质量控制
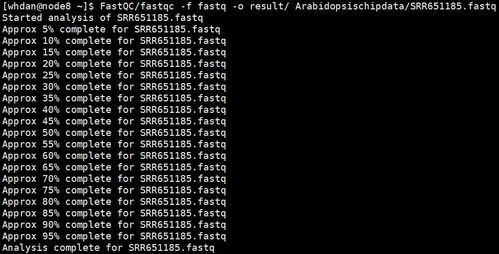
1、质量控制的测序质量检测是通过FastQC软件实现。fastqc可以不设置任何参数运行,这样会直接在当前目录下生成一个质量报告的压缩文件和文件夹,报告是网页格式。也可以设置输出目录和是否解压缩(--noextract),默认设置会解压缩。
2、因为实验过程丌可知,物种特性难量化,数据通过qc,可以做到量化展示数据,从数据分析相关信 息,同时为后续Kmer分析做准备,获取一个准确的基因组预估情况。
3、首先,对二代测序数据进行预处理,去除低质量的序列,去除序列中的重复部分。其次,进行序列比对和变异检测,确定每个序列在基因组上的位置是否正常。最后,对比数据和检测结果,得出最终分析报告。
4、RNA-Seq原始数据质量控制(QC)是非常重要的一个环节,由于各种原因,例如测序平台、实验操作等,原始测序数据可能存在不少问题,如低质量读段、接头序列、污染序列等。为了确保后续分析的准确性,需要先进行质量控制。
3、RNAseq(3)--对RNAseq测序数据的质量控制(fastqc)
1、写在前面 参考1 参考2 1 Trim Galore 是对 FastQC 和 Cutadapt 的包装。2 trim_galore适用于所有高通量测序,包括RRBS(Reduced Representation Bisulfite-Seq ), Illumina、Nextera和smallRNA测序平台的双端和单端数据。
2、RNA-Seq分三个主要的步骤:注意 :我使用 Illumina协议(protocol)和测序仪(sequencer) 作为我的例子,因为他们是常用的,但记住,有其他协议和测序仪是不同的。
3、Illumina提供的mRNA测序技术可在整个mRNA领域进行各种相关研究和新的发现。mRNA测序不对引物或探针进行设计,可自由提供关于转录的客观和权威信息。
4、来源还是 生信技能树 。高通量测序产生的海量数据都是经过压缩再上传的,目前比sra更好的压缩方式也正在研究中。
5、转录组测序是最常用的组学实验,对全谱基因定量,找到差异表达基因。
6、如果这时你还是想获得更多的变异,那么到头来还是得花更多的钱加大测序深度; (3). 目前对转录本数据进行变异检测,还是一个偏于补充性质的分析。 RNA-Seq的目的主要还是集中在基因表达方面,以及寻找差异表达基因和融合基因上。
ChIP-seq数据质控与过滤
1、比如 trim_galore 的这个参数(默认是非常严格:数值1):大部分的ChIP-seq数据都是短读长,去低质量不是必须的。
2、最终的结果表明这6个数据的质量都很好,所以原文并没有进行额外的过滤操作。
3、新手,刚做完一个ChIP-Seq项目的分析,来记录一下,会分好几篇。首先是下机数据fastqc之后会生成一个html格式的报告,根据报告可以看出自己数据的特点,便于之后clean的参数设置。
4、ChIP-Seq的原理是:首先通过染色质免疫共沉淀技术(ChIP)特异性地富集目的蛋白结合的DNA片段,并对其进行纯化与文库构建;然后对富集得到的DNA片段进行高通量测序。
Trim_galore使用(2018-05-25)
1、如果截去接头序列需要在CTGTCTCTTATACACATCT位置截取,工具可以是cutadapt、trim_galore等类似可以自定义截去接头的软件。
RNA-Seq数据分析——原始数据质量控制(QC)
RNA-Seq原始数据质量控制(QC)是非常重要的一个环节,由于各种原因,例如测序平台、实验操作等,原始测序数据可能存在不少问题,如低质量读段、接头序列、污染序列等。为了确保后续分析的准确性,需要先进行质量控制。
使用命令 fastqc -o seqfile1,seqfile. 来进行质量报告。需要注意的是./fastqc前面的.不可以省略 每个fastqc文件会获得一个质量分析报告,来描述此次RNA-seq的测序质量。
RNA-seq数据分析是一个复杂的过程,主要分为以下步骤:数据质量控制:检查原始测序数据的质量,去除低质量的读段(reads)。序列比对:将质量控制后的读段与参考基因组或转录本数据库比对,以确定它们的位置。
该版本没有事先进行质量控制,因此我们可以应用。 我们使用几种常见的QC指标,根据表达谱来鉴定低质量的细胞。
multiqc可以整合其它软件的报告的软件,能将fastqc生成的多个报告整合成一个报告的软件,这样能方便的查看所有测序数据的质量。安装:运行:multiqc可以自动检测到文件中可以整合在一起的文件,运行也很简单。
Seurat是一种R包,设计用于QC,分析和探索单细胞RNA-seq数据。 Seurat旨在使用户能够从单细胞转录组测量中识别和解释异质性来源,并整合不同类型的单细胞数据。
fastqc命令说明的介绍就聊到这里吧,感谢你花时间阅读本站内容,更多关于fastload命令、fastqc命令说明的信息别忘了在本站进行查找喔。
本文章内容与图片均来自网络收集,如有侵权联系删除。
相关推荐
- 资讯排行
- 标签列表
- 友情链接